近日,山东省生物物理重点实验室、生物物理研究院王吉华教授、周百灵副教授带领团队在国际期刊《Briefings in Bioinformatics》(IF=13.994)发表论文,该成果在国家自然科学基金和山东省青创人才引育计划的资助下完成,发挥青创团队多学科交叉优势,与深圳湾实验室资深研究员周耀旗教授、中山大学杨跃东教授合作,使用生物学、信息学、人工智能等多学科交叉研究方法,发展了首个预测潜在功能性长非编码RNA(lncRNAs)的深度学习方法,为lncRNAs的功能研究提供理论依据。相关研究成果以“EVlncRNA-Dpred: Improved prediction of experimentally validated lncRNAs by deep learning”为题发表于国际著名期刊《Briefings in Bioinformatics》(IF=13.994)。
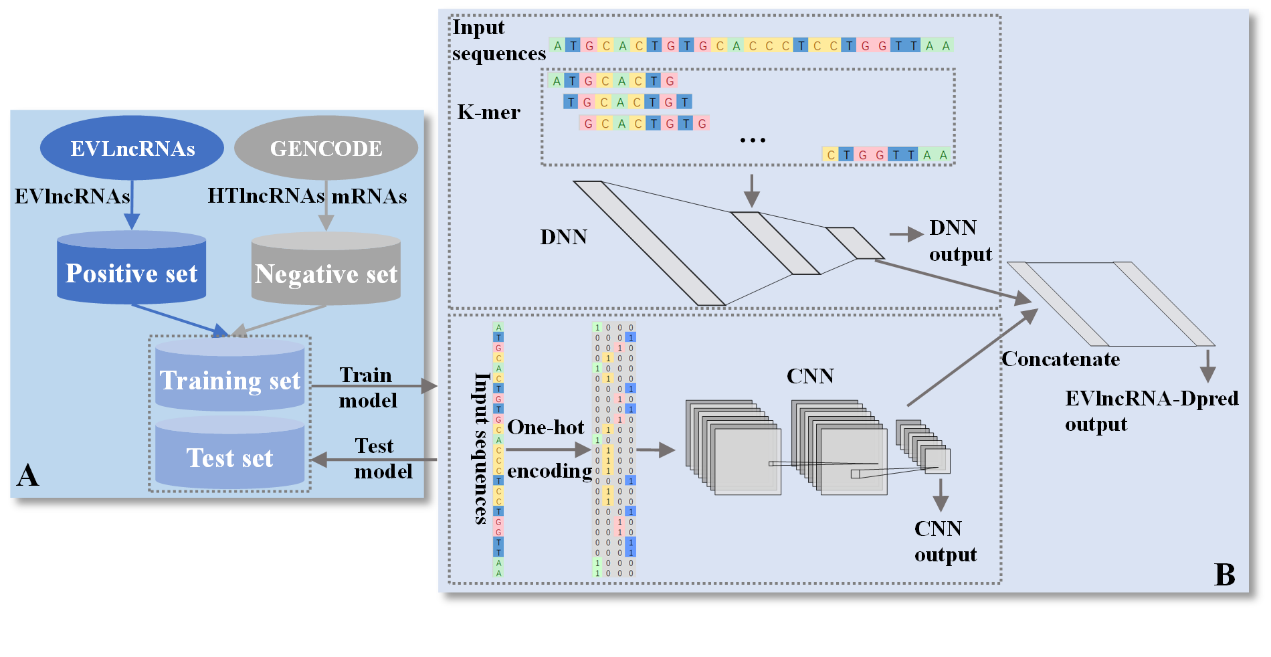
EVlncRNA-Dpred流程示意图
LncRNAs是长度大于200nt、不能编码蛋白质的RNA,在表观遗传修饰、mRNA的转录、剪接、翻译等众多生命过程中发挥着重要生物功能,与人类重大疾病密切相关。科研人员发展了许多生物信息学算法来区分转录组测序数据中的lncRNAs和mRNAs,并结合高通量测序预测发现了60多万个lncRNAs。然而,对lncRNAs进行功能实验研究费时费力,只有一小部分(<1%)的lncRNAs(约4000个)被低通量实验验证(EVLncRNAs2.0,NAR,2021),可谓冰山一角。因此,急需开发生信工具来筛选潜在功能性lncRNAs,为发现新的功能性lncRNA以及揭示其新功能的实验研究提供重要的筛选工具。本研究中,我们采用深度学习方法将实验验证功能性lncRNAs与预测得到的功能未知lncRNAs和mRNAs分离。为了克服小数据集的挑战,我们采用了一个以K-mer特征为输入的三层深度学习神经网络(DNN)和一个以One-hot编码为输入的小型卷积神经网络(CNN),分别训练适用于人、小鼠和植物的三种不同模型。相比之前我们发展的基于支持向量机的模型(EVlncRNA-pred)(RNA Biology, 2019)。本研究获得的EVlncRNA-Dpred模型实现了实质性的改进,有助于筛选潜在功能性lncRNAs,为进一步发现新的功能性lncRNA提供了有力的预测工具,同时,随着实验验证的功能性EVlncRNAs数据的不断丰富扩展,EVlncRNA-Dpred为深入系统发展基于机器学习的功能性lncRNA精准预测方法奠定了基础。为了方便科研人员使用,EVlncRNA-Dpred构建了web服务器,地址为https://www.sdklab-biophysics-dzu.net/EVlncRNA-Dpred/index.html,并支持模型所用数据和源代码的免费获得。
EVlncRNA-Dpred在线预测平台
德州学院为第一完成单位,生物物理研究院周百灵副教授为论文第一作者,王吉华教授和深圳湾实验室周耀旗教授为共同通讯作者。该研究得到了国家自然科学基金、山东省青创人才引育计划、山东省自然科学基金、德州学院人才引进项目等经费资助。此外,本研究还得到中山大学杨跃东教授团队的支持。
多年来,在学校大力支持下,我校山东省生物物理重点实验室研究团队充分发挥省重点实验室研究平台的优势,通过多学科交叉,利用生物信息学、人工智能深度学习等方法解决生命科学的前沿问题,产出了系列创新性成果。下一步,本实验室将在本研究基础上进一步在lncRNAs生物信息学、深度学习算法以及交叉汇聚新技术等方面开展研究,力争实现高水平科研成果进一步突破。
论文链接地址:https://doi.org/10.1093/bib/bbac583