核糖开关不仅可以进行转录调控,还可以在翻译过程进行基因表达调控,能特异性识别或选择配体,是一类在抗菌类药物设计和代谢类生物传感器等方面有广阔应用前景的非编码RNA。
山东省生物物理重点实验室扈国栋教授课题组,采用多种计算和分析方法对腺嘌呤核糖开关的小分子结合机制与其构象变化的关系;PreQ1核糖开关的配体解离结合路径;镁离子对SAM/SAH核糖开关的影响等进行了系统研究。
核糖开关是一种自然存在的RNA适配体,通过与特定的小分子结合来控制细菌基因的表达。高亲和力和高特异性的结合引起构象变化,核糖开关被认为是开发抗生素和化学工具的一个可能的分子靶点。腺嘌呤核糖开关不仅能与嘌呤类似物结合,也能与嘧啶类似物结合。采用长分子动力学(MD)模拟和分子力学Poisson-Boltzmann表面积(MM-PBSA)计算方法,研究结合模型的差异以及配体结合后的构象变化。从结合自由能的角度分析结合能力;寻找与配体结合的关键核苷酸;嘌呤和嘧啶类似物的比较,配体结合引起的动力学特征不同;揭示了构象变化与配体结合的关系。
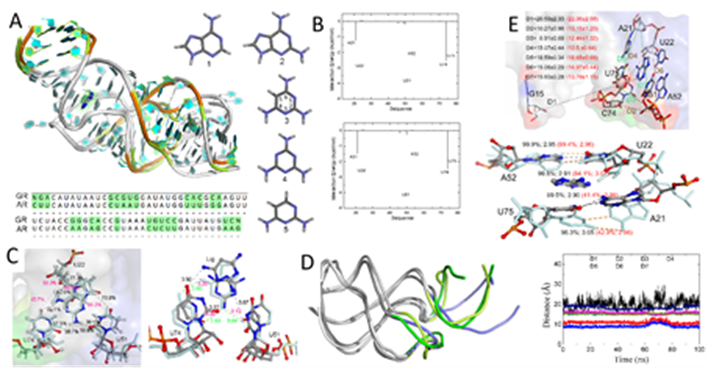
图1. 腺嘌呤小分子的结合机制与其构象变化的关系。(A)鸟嘌呤和腺嘌呤核糖开关结构对比和序列的比较。(B)抑制剂和核苷酸的结合能。(C) 两个典型结构的叠加。(D) 两个典型结构和一个复合物的叠加。(E)结合腔内两个核苷酸的主链原子质量中心之间的距离和构象变化。
采用常规和增强采样MD模拟,共153.5ms,研究了PreQ1核糖开关适配体与同源和合成配体结合的分子机制。通过对配体的结构、能量和动力学性质的分析,我们进一步认识到配体同源和合成基团之间的明显差异以及每一组配体内部的细微差异,特别是氢键供体和受体的减少是合成配体结合亲和力下降的主要原因,而环数的增加导致结合口袋的后门打开。 相对于同源配体Q1小分子,合成配体在结合腔中更容易移动,相应的RNA分子也更灵活。结合自由能分析表明,核苷酸G5、U6、G11、C15、C16和A29通过氢键和碱基堆积作用对同源配体的结合贡献最大。在合成配体的结合中,氢键和碱基堆积都受到了损害,U6、C15和A29不再发挥重要作用。增强采样模拟进一步揭示了同源配体和合成配体以几乎相反的方向从结合腔解离。比较了三种不同的RNA和Mg2+力场组合,验证研究结果的鲁棒性,模拟结果直接验证了实验数据。进一步证明了分子动力学模拟能够补充结构解析,以提供实验缺失的关键环节。提出了一个额外的模式,配体可以通过多种途径进入和退出结合腔,同源配体和合成配体正好相反。可以设计一些实验来进一步测试MD模拟的预测。氢键数目可以通过合成以极性共价键取代C-C或C-H键的L1、L2和L3类似物来验证。Q1形成的可替代氢键,整个L3配体可以有不同的构型, 这些预测可以用核磁共振实验来验证。模拟揭示的同源和合成配体相反的解离和结合路径也可以通过实验进行测试。
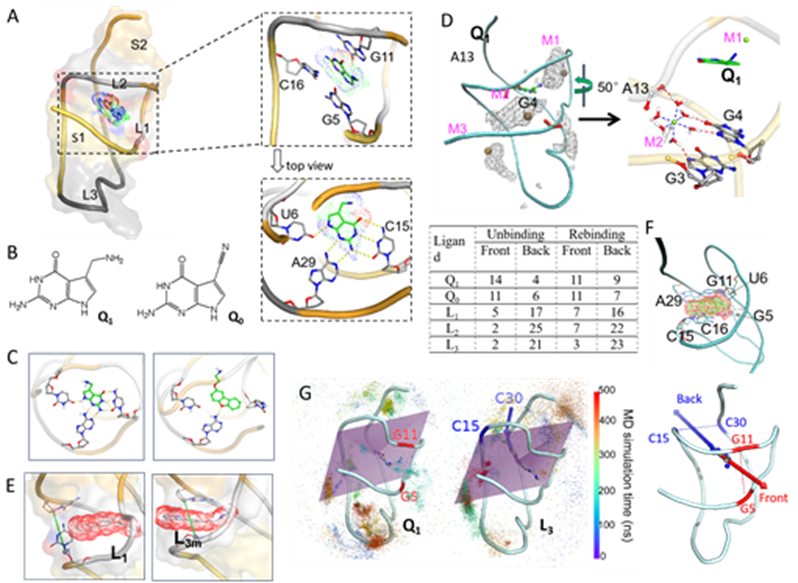
图2. PreQ1核糖开关的配体解离结合路径。(A) Pre Q1-适配体复合物的结构。 (B) Pre Q1和Pre Q0的化学结构。(C)在cMD模拟的典型结构。(D) Q1结合复合物中Mg2+离子的密度轮廓。(E)分别L1-和L3m的典型构象。(F)配体的密度轮廓。(G)小分子的运动轨迹。
SAM/SAH核糖开关以相似的亲和力结合S-腺苷蛋氨酸(SAM)和S-腺苷同型半胱氨酸(SAH)。Mg2+起到稳定RNA结构和中和RNA负电荷的作用。对于SAM/SAH 核糖开关, Mg2+有助于配体结合。研究了Mg2+如何与SAM/SAH核糖开关适配体结合? Mg2+是如何识别; 配体结合对碱基相互作用的影响;配体与Mg 2+结合对RNA构象的协同作用;为什么SAM/SAH核糖开关对SAM和SAH配体都没有选择性?

图3. 镁离子结合到SAM/SAH核糖体。(A) SAH在SAM/SAH riboswitch的结合腔。(B) dU16-G9和dC14-A6,密度图,和结合腔内的示意图。(C) Mg2+与磷酸基相互作用的三种模型。(D) dO4-N1,dM4-G和dM5-G的密度。
我们采用多种计算方法,对核糖开关行驶功能的与小分子以及离子的关系进行了研究。发现配体的化学特征(如氢键供体和受体的数量)和物理特征(如分子大小)影响结合亲和力和配体结合路径;计算结果给出了多个实验可以验证的预测,推动了相关实验的开展;为核糖开关配体的设计提供了新的视角,提出了可行的药物设计策略,有利于新的抗菌药物的设计。 .
本研究得到国家自然科学基金、山东省自然科学基金和美国NIH基金的资助。
Hu Guodong; Huan-Xiang Zhou, Binding free energy decomposition and multiple unbinding paths of buried ligands in a PreQ1 riboswitch. PLoS computational biology 2021, 17 (11), e1009603. https://journals.plos.org/ploscompbiol/article?id=10.1371/journal.pcbi.1009603
Hu Guodong; Haiyan Li; Shicai Xu; Jihua Wang, Ligand Binding Mechanism and Its Relationship with Conformational Changes in Adenine Riboswitch. International journal of molecular sciences 2020, 21 1926. https://www.mdpi.com/1422-0067/21/6/1926
Hu Guodong; Xiu Yu; Yunqiang Bian; Zanxia Cao; Shicai Xu; Liling Zhao; Baohua Ji; Wei Wang; Jihua Wang, Atomistic Analysis of ToxN and ToxI Complex Unbinding Mechanism. International journal of molecular sciences 2018, 19 (11), 3523. https://www.mdpi.com/1422-0067/19/11/3524
Hu Guodong; Aaijing Ma; Jihua Wang, Ligand Selectivity Mechanism and Conformational Changes in Guanine Riboswitch by Molecular Dynamics Simulations and Free Energy Calculations. Journal of chemical information and modeling 2017, 57 (4), 918–928. https://pubs.acs.org/doi/abs/10.1021/acs.jcim.7b00139
Hu Guodong; Huan-Xiang Zhou, Magnesium ions mediate SAM/SAH-riboswitch binding. (prepared)